DRUGAI
Target 2035 是一项全球倡议,旨在到2035年为每个人类蛋白开发一种高效且具选择性的药理调控剂,如化学探针。本文介绍了 Target 2035 为突破小分子初始命中化合物发现这一关键瓶颈而制定的计算策略路线图。研究人员将通过亲和选择质谱(AS-MS)和 DNA 编码化学库筛选,构建大规模、高质量、公开可用的蛋白–小分子结合数据集,涵盖正负样本。机器学习社区将被鼓励利用这些数据构建模型,预测新颖多样的小分子结合物,并通过反复的预测与实验验证迭代优化模型。预计到2030年,该计划将为数千种人类蛋白鉴定出实验验证的初始命中物,并推动开发能够预测尚无实验数据靶点命中物的开放算法。

化学探针——即对特定蛋白具有高效、选择性、可在细胞中活性的微小分子——是生命科学研究中最有影响力的工具之一,已在药物发现和基础研究中发挥了重要作用。为所有人类蛋白质开发化学探针,将极大促进人类蛋白质组的理解,并帮助确定新的潜在药物靶标。
2009 年,结构基因组联盟(SGC)启动了一项计划,专注于为与细胞信号传导、蛋白稳态和表观遗传学相关的人类蛋白开发和收集化学探针。该计划已成功为 200 多种独特蛋白开发了化学探针,并将超过 6 万个样品分发给全球科学家,被引用超过 1.3 万次,并在 85 项以上的临床试验中得到验证。
开发新蛋白化学探针的首要步骤是识别经验证且具有化学可行性的初始命中物。对于已知靶点类别的蛋白,可以通过筛选针对性的化学库或基于已有实验数据的计算预测来获得命中物。相比之下,对于研究较少的蛋白,初始命中物发现难度大且耗时。通常,命中物发现需从大规模化学库的实验筛选开始,并经历耗时、昂贵的验证与优化过程。尽管过去 20 年命中物筛选的实验方法已显著增加,但整体成功率和成本效益提升有限,这凸显了 Target 2035 计划中对全新策略的需求。
计算方法,特别是机器学习(ML)和人工智能(AI),在开发低成本、高效率的命中物发现方法方面潜力巨大。然而,当前公开的蛋白–配体数据集不足以支持此类算法的发展。现有化学活性数据分散在 ChEMBL、PubChem 等数据库中,缺乏标准化实验协议,数据噪声大且难以直接用于 ML/AI 模型训练。此外,大多数数据缺乏关于无活性化合物的高质量信息。
鉴于数据匮乏是命中物发现算法发展的最大障碍,Target 2035 工作组决定在 2025–2030 年阶段组织一项计划:(1)系统生成大规模蛋白–小分子结合数据集,并对高质量注释数据开放访问;(2)与社区合作训练、开发、优化和评估命中物发现算法。
该计划的科学与运营方案(包括靶点选择、数据生成与共享、ML/AI 预测评估、成功标准、治理与资金)已在 2023 年和 2024 年的研讨会上讨论,本文整合了这些成果并提出可行的路线图。
项目工作流程概览
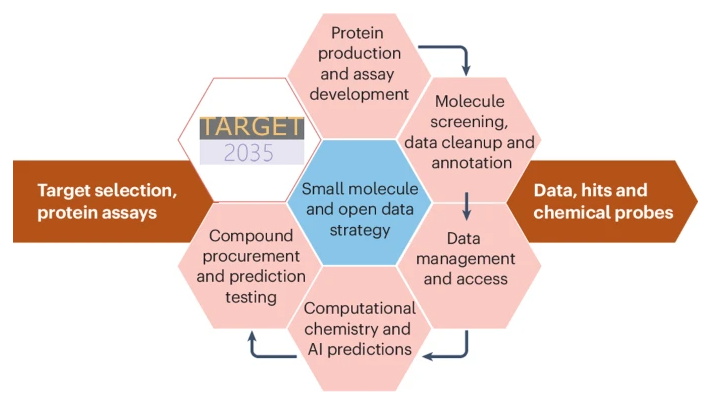
该五年期项目旨在生成包含数百万至数十亿小分子与 2,000 多种多样化蛋白结合数据的高质量开放数据集。这些数据将包括对实验筛选获得的命中化合物及计算预测候选物的验证结果,并通过多种物理和功能测定手段进行验证。
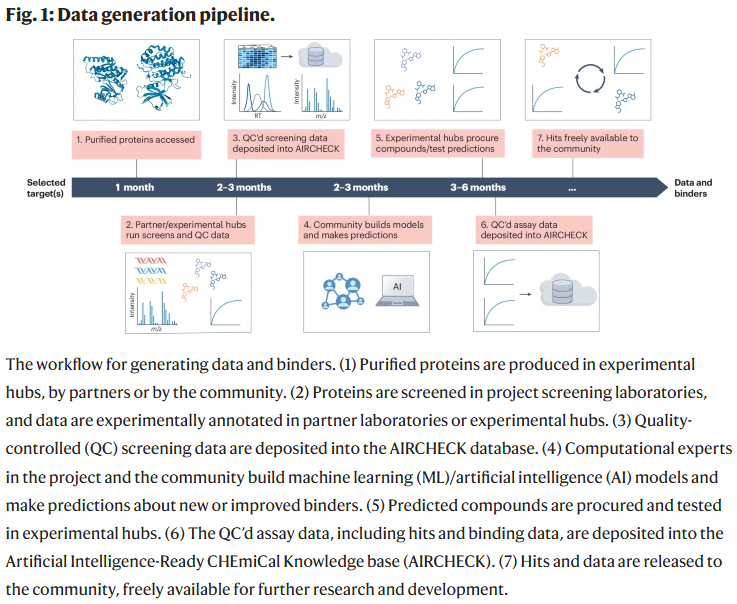
项目流程包括:
蛋白生产:在项目内部或通过社区贡献生成纯化蛋白,并进行严格质量控制。
结合数据生成:使用亲和选择质谱(AS–MS)和 DNA 编码化学库(DEL)筛选来测定小分子与蛋白的直接结合情况,随后用正交的高质量生物物理测定进行验证。
开放数据发布:将带注释的初级筛选数据以 ML/AI 可用格式存入 AIRCHECK 数据库。
模型挑战与竞赛:鼓励 ML/AI 和计算化学领域基于这些数据进行预测,并通过基准挑战推动方法改进。
实验验证:使用生物物理手段验证社区预测结果。
数据共享:将验证数据及所有试剂、协议、结合物和数据无条件地公开共享。
此外,将尽可能尝试蛋白–配体复合物的共结晶,并探索已确认结合物的结构–活性关系研究。
获取多样化高质量蛋白
为生成足够规模和多样性的蛋白–小分子结合数据集,必须获得结构多样且稳定的纯化蛋白。项目计划在 5 年内筛选至少 2,000 种蛋白,这与 SGC 在 2007–2012 年期间生产的独特蛋白数量相当,具备可行性。
蛋白的选择将基于结构和功能多样性,并考虑资助方与参与者的兴趣。初期将优先选择实验可行性高的靶点,以建立和优化平台与数据流程。随着项目推进,将逐渐纳入更具挑战性的蛋白。
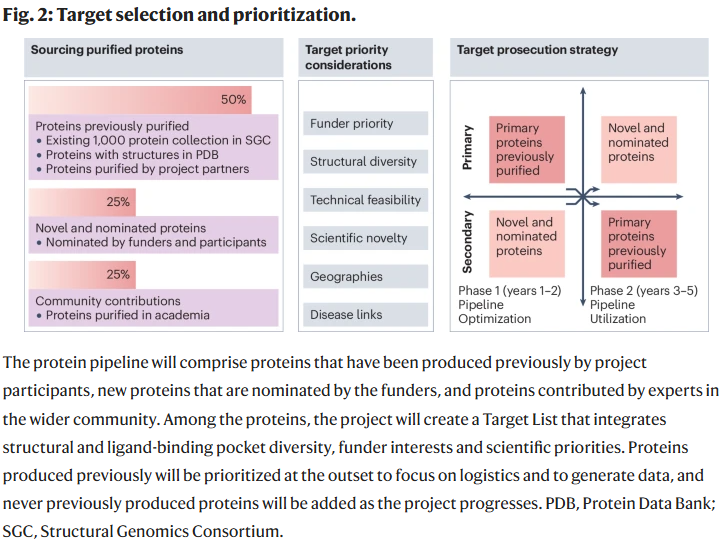
蛋白生产
项目制定了严格的蛋白质量标准,大部分蛋白将在分布于不同地区的纯化中心生产,这些中心共享方法与标准。社区专家可提交符合标准的蛋白,以获得高质量化学筛选服务及后续命中化合物的研究权利。
蛋白–配体开放数据生成
通过高质量的 AS–MS 和 DEL 筛选生成结合数据。选择这两种平台的原因包括:
直接结合测定避免了对功能测定的依赖。
同一批纯化蛋白可用于初级筛选和正交验证。
目前已纯化的数百种人类蛋白可立即用于筛选。
最终生成的数据可整合为标准化、可被 ML 直接利用的大型数据集。
DEL 筛选
DEL 筛选是一种基于亲和选择的技术,利用连接 DNA 条码的小分子库与蛋白孵育,然后通过捕获和洗涤分离结合物。DNA 条码序列的测序可揭示每个小分子的合成历史和富集程度。
与 ML/AI 相结合,DEL 可通过海量正负样本数据训练算法,预测潜在结合物。这样研究人员可直接从商业化学库中筛选候选物并通过实验验证,大大减少了传统 DEL 富集物的重新合成工作量。
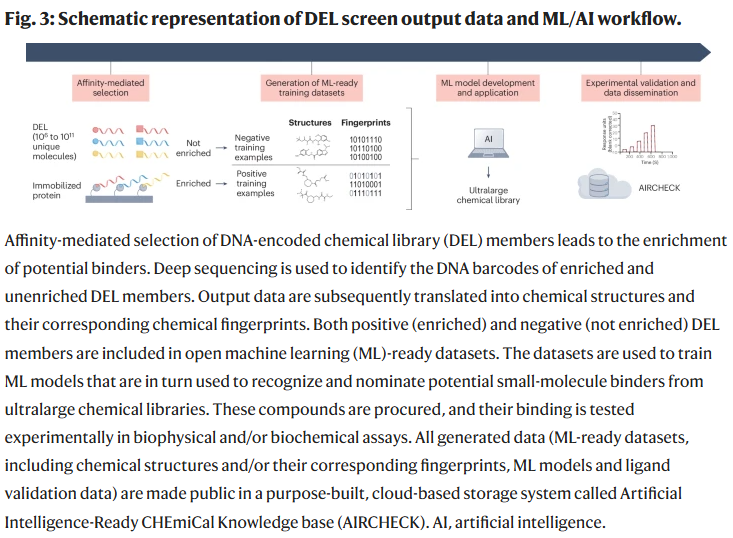
AS–MS 筛选
AS–MS 是制药行业已验证的命中物识别技术。通过将多达 2,000 种质量区分化合物与目标蛋白共孵育,利用液相色谱–质谱分析确定共洗脱的化合物并验证其结合能力。该技术对低微摩尔级结合物的检测十分有效,且能与 DEL 筛选互补。
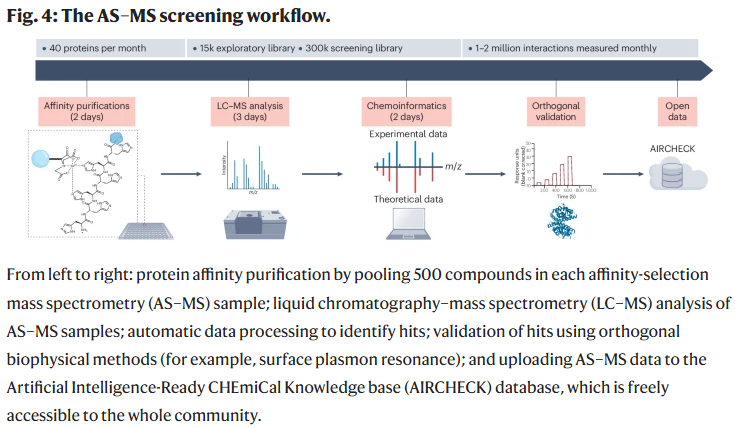
筛选数据注释与验证
为保证数据适用于 ML/AI,研究人员对数据质量、注释与可用性实施严格标准。包括:
蛋白质:确保纯化条件及其他关键元数据齐全。
初级数据:AS–MS 和 DEL 数据需通过技术质量检查后开放。
正交验证:通过多种物理测定区分真伪阳性,并确定结合物的亲和力阈值(如 KD ≤ 10 μM)。
数据管理与访问
数据管理遵循 FAIR 原则,采用自动化实验记录、集中式数据库架构,并提供透明可复现的处理流程。AIRCHECK 平台将实现数据发布、版本控制、模型共享和不间断的预测性能监控。
基准测试与实验反馈
项目计划通过定期组织社区预测挑战(类似 CASP、DREAM 竞赛)推动命中物预测算法的发展。预测结果将通过实验验证并与真实数据对比,以确保模型的可靠性。
从试点到全面实施
项目试点阶段已完成核心能力建设,包括蛋白生产、筛选、数据存储和注释。正式实施阶段将整合这些元素,实现命中物识别与长期数据积累的平衡。
促进社区贡献
该项目坚持开放科学原则,所有化合物、数据和算法将无专利限制地开放使用。社区贡献者可以提供蛋白、化合物或预测模型,项目则免费提供筛选数据与验证结果作为回报。
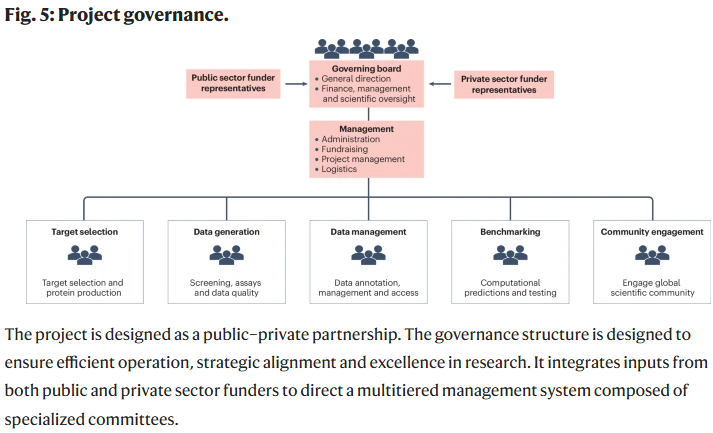
项目结构与治理
项目采用公共–私营合作模式,治理结构包括公共和私营资助方的联合董事会,以保证资金、科学与运营的平衡。所有生成的数据与算法在署名后都可自由使用。
讨论
项目的长期目标是开发高效的计算命中物发现算法,为数千种乃至所有相关人类蛋白生成可自由获取的小分子结合物。过程中产生的中间成果同样具有重要价值,可作为项目进展的度量指标,如蛋白数量、筛选次数、命中物新颖性、ML 模型性能提升和社区参与度等。
开放获取的公共–私营合作模式能最大限度整合跨学科力量,消除知识产权障碍并促进全球科研协作。对学术界而言,项目提供了真实世界的高质量数据和跨学科训练机会;对工业界和政府部门而言,项目推动了新型筛选技术、ML 模型和潜在药物起点的开发;对于基金会和社会,项目为实现早期药物发现民主化提供了平台。
最终,研究人员相信,通过大规模高质量蛋白–配体数据集与开放式 ML/AI 预测模型的结合,Target 2035 能够大幅加速命中物发现的计算化转型,并为未来药物研发和生命科学研究提供坚实的基础。
整理 | WJM
参考资料
Edwards, A.M., Owen, D.R. & The Structural Genomics Consortium Target 2035 Working Group. Protein–ligand data at scale to support machine learning. Nat Rev Chem (2025).
https://doi.org/10.1038/s41570-025-00737-z
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢